FUNKTIONELLE PROTEOMIK – Neurodegenerative Erkrankungen
Funktionelle Proteomik
Proteine als Schlüssel für die Diagnostik
Im Kompetenzbereich Medizinische Proteomanalyse liegt ein Forschungsschwerpunkt auf der Identifizierung sowie Validierung von Proteinveränderungen, die im Zusammenhang mit neurodegenerativen Erkrankungen auftreten. Dafür entwickeln wir unter anderem hoch-sensitive Massenspektrometrie-basierte Ansätze für die Proteom- und Proteinanalytik. Für die prognostische und prädiktive Diagnostik ermitteln wir Protein-Signaturen, die potentiell als krankheitsrelevante Biomarker verwendet werden können. Für die Identifizierung von Biomarkern verwenden wir Rückenmarksflüssigkeit und Plasma. Zudem fokussieren wir uns auf die Erforschung der zugrundeliegenden Krankheitsmechanismen. Mittels spezifischer Gewebe-Analysen, bei denen wir einen kombinierten Ansatz der Lasermikrodissektion-Massenspektrometrie anwenden, werden molekulare Prozesse auf Proteinebene identifiziert, die fundamentale Erkenntnisse für diagnostische und therapeutische Ansätze bieten können.
Protein-Signaturen für die Diagnostik
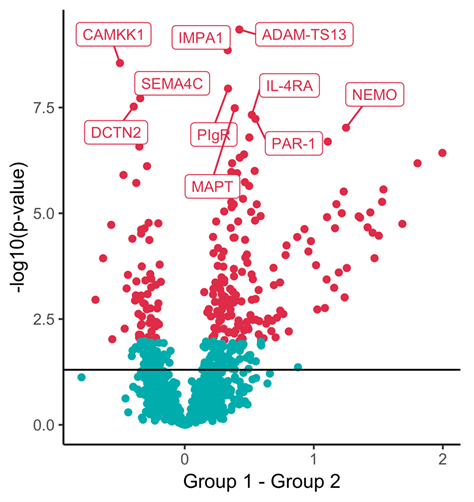
Neurodegenerative Erkrankungen, wie Alzheimer und Parkinson, sind durch einen fortschreitenden Verlust von Nervenzellen sowie das Fehlen einer adäquaten Diagnose und Behandlung gekennzeichnet. Viele neurodegenerative Erkrankungen werden als Proteinopathien beschrieben und sind durch die Ansammlung von krankheitsspezifischen Proteinaggregaten im Gehirn gekennzeichnet. Für die Diagnostik spielt Rückenmarksflüssigkeit/Liquor, die in direktem Kontakt mit dem zentralen Nervensystem steht, eine zentrale Rolle, denn Änderungen im Gehirn können zu u.a. abnormaler Proteinabundanz oder veränderten Proteinmodifizierungen im Liquor führen. Die betroffenen Proteine repräsentieren potenzielle Biomarker, die für die Diagnostik genutzt werden können. Bekannte Krankheits-assoziierte Proteine sind z. B. Amyloid-β Peptide, Tau oder Alpha-Synuklein. Zur Identifizierung als auch Quantifizierung von Biomarkern nutzen wir hochauflösende Massenspektrometrie. Durch dieses Verfahren können wir in differenziellen klinischen Studien (Vergleich gesund gegenüber krank) Krankheits-spezifische Protein-Signaturen auf globaler Ebene identifizieren. Erste vorläufige Protein-Biomarker-Signaturen konnten wir im Liquor bereits für die Frühdiagnose der Parkinson-Krankheit finden. Eine Herausforderung bei der Identifizierung von Biomarkern ist das jeweilige Ausmaß der Veränderung. Der Grad der krankheitsbedingten, abnormalen Proteinabundanz ist zu Beginn einer Erkrankung sehr gering und kann unter Umständen aufgrund der Sensitivität und Spezifität der Analyse-Technik kaum messbar sein. Dafür entwickeln wir spezifische und hoch-sensitive zielgerichtete Massenspektrometrie-basierte Ansätze (z.B. Barkovits et al. J Proteome Res. 2018; Barkovits et al. Methods Mol Biol. 2019; Hörmann et al. Methods Mol Biol. 2019; Barkovits et al. Cells 2020; Barkovits et al. Mol Cell Proteomics 2020).
Aufdeckung von Neurodegenerationsmechanismen
Bei neurodegenerativen Erkrankungen sind in erster Linie Nervenzellen des menschlichen Nervensystems betroffen. Nervenzellen sind hochspezialisierte Zellen, die für die Informationsübertragung und -verarbeitung im Gehirn zuständig sind. Im Vergleich zu anderen Zelltypen können sich Nervenzellen grundsätzlich nicht selbst ersetzen, daher können abgestorbene Nervenzellen vom Körper nicht wieder erneuert werden. Bei Erkrankungen des Nervensystems kommt es meist zu einem Verlust von Nervenzellen im bestimmten Gehirnbereichen. Bei der Parkinson-Krankheit beispielsweise degenerieren Nervenzellen selektiv in der Substantia nigra pars compacta, während bei der Alzheimer-Krankheit eine globale Degeneration der Nervenzellen im Hippocampus und in der Großhirnrinde stattfindet. Abhängig von dem betroffenen Bereich führt der Verlust von Nervenzellen zu einer Beeinträchtigung der Bewegungsabläufe oder der mentalen Fähigkeiten bis hin zum Tod. Um aufzuklären, wie sich das betroffene Gewebe vom gesundem unterscheidet und warum bei verschiedenen neurodegenerativen Erkrankungen unterschiedliche Bereiche im Gehirn betroffen sind, nutzen wir einen Lasermikrodissektion-basierte Ansatz. Die Lasermikrodissektion ermöglicht die Isolierung spezifischer zellulärer sowie subzellulärer Bereiche des Gehirns und erlaubt uns somit eine gezielte Untersuchung der von der Krankheit betroffenen Strukturen. Durch die Kopplung der Lasermikrodissektion mit der hoch-sensitiven Massenspektrometrie können wir spezifische Proteomprofile erstellen und somit einen globalen Überblick über Prozesse erhalten, die normalen/gesunden sowie Krankheits-bedingt veränderten zellulären Abläufen auf Proteinebene zugrunde liegen. So ist es uns bereits gelungen, krankheitsspezifische Veränderungen im Proteom einzelner Neuronenpopulationen (Steinbach et al., in Vorbereitung) und des Pigments Neuromelanin (Plum et al., 2016) zu identifizieren.